RNA seq 분석을 하다보면 Heatmap을 그려야 할 상황이 올때가 있다.
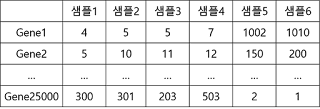
아래의 예시 그림은 6개 샘플에 대한 유전자 heatmap이다. R 코드 세줄이면 아래 그림과 같은 멋있는 heatmap을 그릴 수 있다.
먼저 인풋 파일은 RNA-Seq 파이프라인에서 FeatureCounts등의 툴을 돌려 정량화(quantification)단계 후 EdgeR이나 DESeq을 통해서 Normalization된 샘플별 유전자 발현량(EdgeR의 경우 TMM)을 나타낸 파일이 있으면 된다.
<GeneExpresson.txt>
Exp <- read.table("GeneExpresson.txt",sep="\t") #샘플 파일 열어주기library(pheatmap) #pheatmap 패키지 열어주기. 없으면 설치할것!pdf("Heatmap.pdf", width =7, height=14, onefile=FALSE) #pdf로 저장하기위해서 빈페이지를 열어줌pheatmap(Exp,cluster_rows = T , fontsize_row = 3.5,cluster_cols = T)dev.off() #pdf를 닫아줌.
위에 코드 세줄만 입력하면 위의 그림과 같은 heatmap이 나온다! 코드 자체는 어렵지 않으나 여기서 알아야할 이슈 몇가지를 소개해 보고자 한다.
1. 만약 normalization 된 발현량 값이 당장 없을 경우 어떻게 하나요? 이때는 pheatmap 안에 있는 scale 옵션을 이용하여 일단 RNA-seq 데이터에는 적합하지 않지만 임의로 normalization을해주는 방법이 있다.
pheatmap(Exp,cluster_rows = T , fontsize_row = 3.5,cluster_cols = T, scale ="row")
2. 위에 Cluster 옵션(cluster_rows = T)은 뭔가요?
만약 저 옵션을 True로 해준다면 비슷한 값의 패턴을 가진 샘플끼리 행순서(여기서는 유전자 순서)가 다시 재배치되면서 그림이 그려진다. 만약에 row대신에 cols 를 써주면 열순서(여기서는 샘플순서)가 다시 재배치되면서 그림이 그려진다,
아래의 비디오는 heatmap을 그리기 전에 normalization 된 값(TMM noramlization)을 어떻게 구하는지에 대한 영상입니다.